Alle Organismen sind in der Lage, wichtige zelluläre Prozesse an den Stoffwechselzustand anzupassen. So wird exakt koordiniert, dass bestimmte Reaktionen, wie die Herstellung von Proteinen und anderer Biomoleküle, nur dann stattfindet, wenn die Zellen nicht unter Nährstoffknappheit leiden. Dazu nutzen diese Organismen Moleküle, die als Indikatoren des Stoffwechselzustandes fungieren und die zudem dazu genutzt werden können, um die Aktivität von Biomolekülen entsprechend daran anzupassen.
Eines dieser zentralen Stoffwechselmoleküle ist die aktivierte Essigsäure, das sogenannte Acetyl-Coenzym A. Zelluläre Level von Acetyl-CoA können genutzt werden, um den Stoffwechselzustand der Zellen zu messen. Ist die Konzentration in den Zellen hoch, deutet das auf eine hohe Stoffwechselaktivität unter Bedingungen einer reichlichen Verfügbarkeit von Nährstoffen hin. Ist die Konzentration hingegen niedrig, liegen Bedingungen der Nährstoffknappheit vor. Frühere Studien haben gezeigt, dass Acetyl-CoA in Zellen enzymatisch und nicht-enzymatisch zur Acetylierung von Biomolekülen führt. Damit die Modifikation effizient und gezielt von diesen Biomolekülen wieder entfernt werden kann, um den Ausgangszustand wieder herzustellen und nicht dauerhaft ein reichhaltiges Nährstoffangebot anzuzeigen, gibt es Enzyme, die sogenannten Deacetylasen.
Eine weitere wichtige Erkenntnis ist zudem, dass diese bakteriellen Enzyme auch in der Lage sind, neben der Acetylierung auch andere Acylierungen von der Aminosäure Lysin in Proteinen zu entfernen. Dieses sind allesamt Modifikationen, die den Stoffwechselzustand der Zellen anzeigen und zudem weitere Funktionen in Bakterien offenbaren könnten, wie eine Membranbindung von Proteinen in den Zellen. Während die Klasse der bakteriellen Zn2+-abhängigen Deacylasen in Eukaryoten wie Tieren und Pflanzen schon recht gut untersucht sind, gab es bisher keine systemischen Studien zu dieser Enzymklasse in Bakterien. Diese Lücke wird mit der aktuellen Studie geschlossen.
„In unserer Studie konnten wir insgesamt zwölf Proteinstrukturen als Repräsentanten der fünf Hauptgruppen dieser bakteriellen Enzyme aufklären. Zusammen mit Funktionsuntersuchungen zu den Aktivitäten und zur Hemmung dieser Enzyme haben wir damit einen sehr guten Überblick über die Struktur und Funktion der Enzyme in Bakterien erhalten“, erklärt die Doktorandin und Erstautorin der Studie Leonie Graf. Professor Michael Lammers, Leiter der Gruppe Synthetische und strukturelle Biochemie, fügt hinzu: „Wir haben selbst nicht erwartet, dass wir so viele bisher nicht beschriebene Kandidaten dieser Enzymklasse in Bakterien finden. Dieses Projekt wird die Grundlage für viele weitere Studien sein, die zeigen werden, welche physiologischen Prozesse über diese Enzyme reguliert werden und ob wir diese Enzyme auch therapeutisch ansteuern können. Auch hier war es die exzellente Zusammenarbeit mit den Arbeitsgruppen von Professor Kay Hofmann von der Universität zu Köln, Professor Richard Strugnell von der Universität Melbourne und Professor Christian Olsen von der Universität Kopenhagen, die uns erst ermöglicht haben, ein so umfassendes Bild dieser Enzymklasse zu erhalten.“
Beteiligte Gruppen
Prof. Michael Lammers, Institut für Biochemie
Prof. Kay Hofmann, Universität zu Köln
Prof. Christian A. Olsen, Universität Kopenhagen
Prof. Richard A. Strugnell, Universität Melbourne
Weitere Informationen
Arbeitsgruppe Prof. Dr. Michael Lammers
Zum Artikel in der Fachzeitschrift Nature Communications (DOI 10.1038/s41467-024-53903-0)
Diese Medieninformation hat die Kurz-URL https://ugreif.de/afkt7.
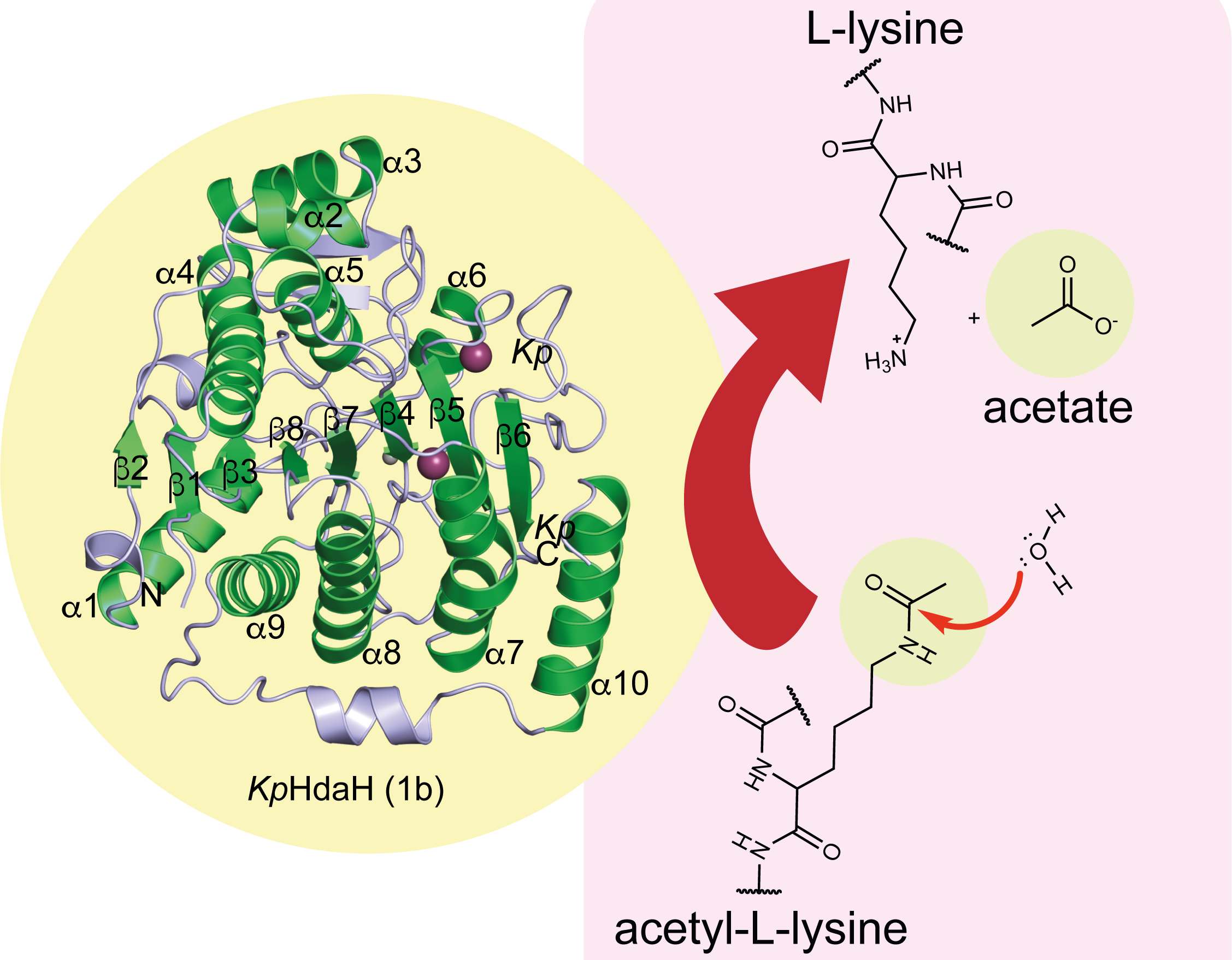
Abbildung
Viele Bakterienarten haben eine Reihe von Enzymen, die in der Lage sind, die Aminosäure oder andere Moleküle zu deacetylieren, wie das hier gezeigte Enzym HdaH aus Klebsiella pneumoniae. © Michael Lammers
Ansprechpartner an der Universität Greifswald
Prof. Dr. Michael Lammers
Institut für Biochemie
Felix-Hausdorff-Straße 4, 17489 Greifswald
Telefon +49 3834 420 4356
michael.lammersuni-greifswaldde